WGCNA怎么挖掘潛在的共表達(dá)基因
這篇文章主要介紹“WGCNA怎么挖掘潛在的共表達(dá)基因”,在日常操作中,相信很多人在WGCNA怎么挖掘潛在的共表達(dá)基因問(wèn)題上存在疑惑,小編查閱了各式資料,整理出簡(jiǎn)單好用的操作方法,希望對(duì)大家解答”WGCNA怎么挖掘潛在的共表達(dá)基因”的疑惑有所幫助!接下來(lái),請(qǐng)跟著小編一起來(lái)學(xué)習(xí)吧!
成都創(chuàng)新互聯(lián)專業(yè)為企業(yè)提供天元網(wǎng)站建設(shè)、天元做網(wǎng)站、天元網(wǎng)站設(shè)計(jì)、天元網(wǎng)站制作等企業(yè)網(wǎng)站建設(shè)、網(wǎng)頁(yè)設(shè)計(jì)與制作、天元企業(yè)網(wǎng)站模板建站服務(wù),10年天元做網(wǎng)站經(jīng)驗(yàn),不只是建網(wǎng)站,更提供有價(jià)值的思路和整體網(wǎng)絡(luò)服務(wù)。
共表達(dá)基因指的是表達(dá)量具有協(xié)同變化趨勢(shì)的基因集合,通常認(rèn)為這些基因參與相同的生物學(xué)過(guò)程,比如參與同一個(gè)代謝通路,正是由于功能上的協(xié)同作用,導(dǎo)致表達(dá)量呈現(xiàn)出高度相關(guān)性。
在WGCNA中,對(duì)傳統(tǒng)的相關(guān)系數(shù)進(jìn)行乘方運(yùn)算,用最終得到的值來(lái)表征基因間的相關(guān)性。在計(jì)算出這樣的相關(guān)性統(tǒng)計(jì)量值之后,如何確定哪些基因是共表達(dá)的呢?
WGCNA的做法是聚類分析,聚類分析屬于一種非監(jiān)督的機(jī)器學(xué)習(xí)算法,通過(guò)聚類樹(shù),可以觀察到哪些基因在聚類樹(shù)中屬于同一分支,屬于同一分支的基因可以歸為一類。實(shí)際操作中,考慮到基因數(shù)目較多等情況,肯定需要算法來(lái)自動(dòng)化的進(jìn)行分類,WGCNA采用的是dynamicTreeCut這個(gè)R包。
對(duì)于聚類算法而言,需要輸入基因間的距離矩陣,首先就需要將基因間的鄰接矩陣轉(zhuǎn)換為距離矩陣,對(duì)相關(guān)系數(shù)進(jìn)行乘方運(yùn)算,可以計(jì)算出鄰接矩陣,但是這個(gè)值本質(zhì)上反映的是基因間的相似度,并不是距離。在計(jì)算距離矩陣時(shí),WGCNA采用了TOM這種統(tǒng)計(jì)量,該統(tǒng)計(jì)量可以表征網(wǎng)絡(luò)中節(jié)點(diǎn)的相似性,計(jì)算公式如下
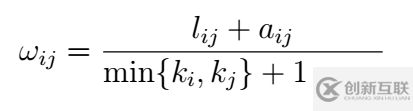
對(duì)于兩個(gè)基因i和j而言,a表示兩個(gè)基因鄰接矩陣中對(duì)應(yīng)的值,就是相關(guān)系數(shù)的乘方,K代表的每個(gè)基因的連接度, 公式如下
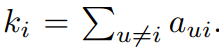
對(duì)于加權(quán)網(wǎng)絡(luò)而言,就是該節(jié)點(diǎn)的邊對(duì)應(yīng)數(shù)值的總和,比如在網(wǎng)絡(luò)中基因A與3個(gè)基因相連,基因A的連接度就是對(duì)應(yīng)3條邊的數(shù)值之和。兩個(gè)基因間的l值代表的是兩個(gè)基因所有邊的權(quán)重乘積的總和,公式如下

公式只是幫助我們理解計(jì)算的過(guò)程,其實(shí)只需要理解TOM是表征節(jié)點(diǎn)的相似度就行,我們要的是距離,所以直接用1減去相似度即可,公式如下

借助TOM值,將基因間的相關(guān)系數(shù)轉(zhuǎn)換為了距離,然后就可以用該距離矩陣進(jìn)行聚類。上述的計(jì)算方法在WGCNA中都有對(duì)應(yīng)的公式,代碼如下
# 確定乘方運(yùn)算中power的最佳取值 powers <- c(c(1:10), seq(from = 12, to=20, by=2)) sft <- pickSoftThreshold( datExpr, powerVector = powers, verbose = 5) softPower <- sft$powerEstimate # 計(jì)算鄰接矩陣 adjacency <- adjacency(datExpr, power = softPower) # 計(jì)算TOM相似度矩陣 TOM <- TOMsimilarity(adjacency) # 計(jì)算距離矩陣 dissTOM <- 1-TOM # 聚類 geneTree <- hclust(as.dist(dissTOM), method = "average")
根據(jù)聚類結(jié)果和距離矩陣,就可以調(diào)用dynamicTreeCut的算法來(lái)識(shí)別modules, 代碼如下
# 指定每個(gè)module中基因數(shù)目的最小值 minModuleSize <- 30 # 識(shí)別modules dynamicMods <- cutreeDynamic( dendro = geneTree, distM = dissTOM, deepSplit = 2, pamRespectsDendro = FALSE, minClusterSize = minModuleSize)
通過(guò)table函數(shù)可以查看modules的結(jié)果,用法如下
> table(dynamicMods) dynamicMods 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 88 614 316 311 257 235 225 212 158 153 121 106 102 100 94 91 78 76 65 58 20 21 22 58 48 34
可以看到,識(shí)別出22個(gè)modules, 0代表那些沒(méi)有歸入任何modules的基因。通過(guò)plotDendroAndColors函數(shù)可視化聚類樹(shù)對(duì)應(yīng)的modules, 代碼如下
dynamicColors = labels2colors(dynamicMods) plotDendroAndColors( geneTree, dynamicColors, "Dynamic Tree Cut", dendroLabels = FALSE, hang = 0.03, addGuide = TRUE, guideHang = 0.05, main = "Gene dendrogram and module colors")
生成的圖片如下

整個(gè)圖片分為兩個(gè)部分,上方為基因的聚類樹(shù),下方為識(shí)別到的modules, 不同的modules對(duì)應(yīng)不同的顏色,其中灰色對(duì)應(yīng)那些沒(méi)有歸入任何modules的基因。
通過(guò)dynamicTreeCut識(shí)別到modules之后,還會(huì)結(jié)合每個(gè)modules的基因表達(dá)量數(shù)據(jù),來(lái)識(shí)別相關(guān)性很高的modules, 從而進(jìn)行合并,其原理是對(duì)modules進(jìn)行聚類,每個(gè)module下的基因表達(dá)量是一個(gè)二維矩陣,做相關(guān)性分析我們只需要一個(gè)一維向量就可以了,可以利用PCA分析提取第一主成分來(lái)表征原始的矩陣,在WGCNA中,把每個(gè)module的表達(dá)譜數(shù)據(jù)對(duì)應(yīng)的一維向量稱之為Module eigengene E。獲取一維向量之后,就可以計(jì)算相關(guān)性,直接用1減去相關(guān)性作為距離,進(jìn)行聚類,代碼如下
MEList <- moduleEigengenes( datExpr, colors = dynamicColors) MEs <- MEList$eigengenes MEDiss <- 1-cor(MEs) METree <- hclust(as.dist(MEDiss), method = "average") plot(METree, main = "Clustering of module eigengenes", xlab = "", sub = "")
modules的聚類樹(shù)示意如下
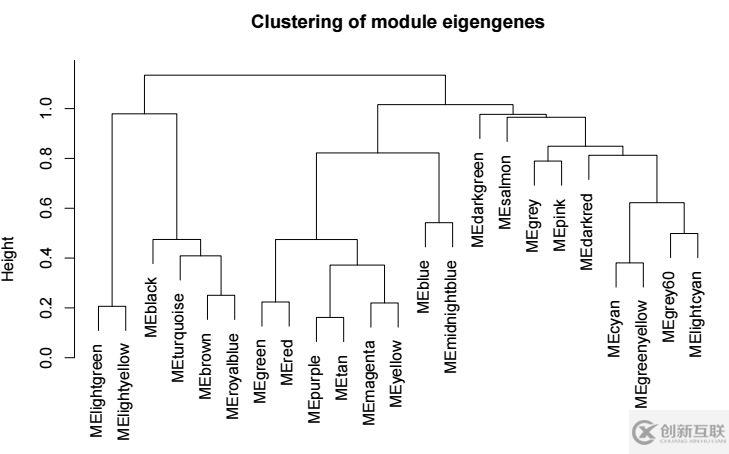
每個(gè)modules的名字用對(duì)應(yīng)的顏色表示,在該聚類數(shù)中,分支長(zhǎng)度為1減去兩個(gè)module間的相關(guān)系數(shù),在合并modules時(shí),將高相關(guān)性的合并為一類,可以指定一個(gè)閾值,比如將相關(guān)系數(shù)大于0.8的合并為一類,在該聚類樹(shù)中,對(duì)應(yīng)的就是height小于0.2的modules, 對(duì)應(yīng)下圖紅色的線
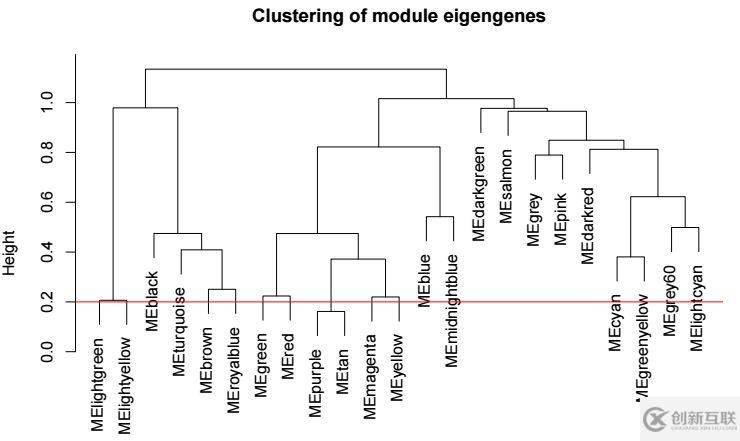
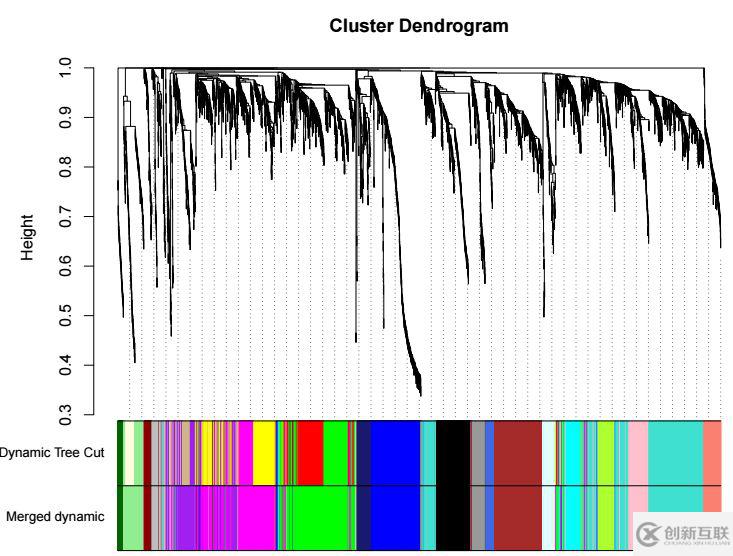
可以看到有8個(gè)modules都滿足條件,在合并時(shí),會(huì)將原本屬于同一分支的modules直接合并為一個(gè),從圖上可以看出,合并后會(huì)減少4個(gè)modules。合并的代碼如下
MEDissThres <- 0.2
merge <- mergeCloseModules(
datExpr,
dynamicColors,
cutHeight = MEDissThres,
verbose = 3)
mergedColors <- merge$colors
mergedMEs <- merge$newMEs
plotDendroAndColors(
geneTree,
cbind(dynamicColors, mergedColors),
c("Dynamic Tree Cut", "Merged dynamic"),
dendroLabels = FALSE,
hang = 0.03,
addGuide = TRUE,
guideHang = 0.05)合并之后的modules 對(duì)應(yīng)的圖片如下
到此,關(guān)于“WGCNA怎么挖掘潛在的共表達(dá)基因”的學(xué)習(xí)就結(jié)束了,希望能夠解決大家的疑惑。理論與實(shí)踐的搭配能更好的幫助大家學(xué)習(xí),快去試試吧!若想繼續(xù)學(xué)習(xí)更多相關(guān)知識(shí),請(qǐng)繼續(xù)關(guān)注創(chuàng)新互聯(lián)網(wǎng)站,小編會(huì)繼續(xù)努力為大家?guī)?lái)更多實(shí)用的文章!
新聞名稱:WGCNA怎么挖掘潛在的共表達(dá)基因
文章出自:http://www.chinadenli.net/article18/iphgdp.html
成都網(wǎng)站建設(shè)公司_創(chuàng)新互聯(lián),為您提供App開(kāi)發(fā)、網(wǎng)站收錄、用戶體驗(yàn)、App設(shè)計(jì)、靜態(tài)網(wǎng)站、商城網(wǎng)站
聲明:本網(wǎng)站發(fā)布的內(nèi)容(圖片、視頻和文字)以用戶投稿、用戶轉(zhuǎn)載內(nèi)容為主,如果涉及侵權(quán)請(qǐng)盡快告知,我們將會(huì)在第一時(shí)間刪除。文章觀點(diǎn)不代表本網(wǎng)站立場(chǎng),如需處理請(qǐng)聯(lián)系客服。電話:028-86922220;郵箱:631063699@qq.com。內(nèi)容未經(jīng)允許不得轉(zhuǎn)載,或轉(zhuǎn)載時(shí)需注明來(lái)源: 創(chuàng)新互聯(lián)

- 要怎么做好品牌網(wǎng)站建設(shè)? 2022-12-28
- 品牌網(wǎng)站建設(shè)的方案 2021-06-13
- 品牌網(wǎng)站建設(shè)價(jià)格雜亂無(wú)章如何定義 2022-10-01
- 品牌網(wǎng)站建設(shè)要注意哪些方面 2020-12-18
- 品牌網(wǎng)站建設(shè)如何傳遞客戶的品牌價(jià)值 2022-08-11
- 品牌網(wǎng)站建設(shè)這六大問(wèn)題您重視了嗎 2016-11-07
- 高端品牌網(wǎng)站建設(shè)該怎么做? 2022-03-10
- 品牌網(wǎng)站建設(shè)只需幾招便可搞定 2014-07-02
- 品牌網(wǎng)站建設(shè)方案要怎么做? 2016-09-10
- 品牌網(wǎng)站建設(shè)有什么作用和優(yōu)勢(shì)嗎? 2022-07-16
- 品牌網(wǎng)站建設(shè)的幾大要點(diǎn) 2023-05-01
- 請(qǐng)相信為你服務(wù)的品牌網(wǎng)站建設(shè)公司專業(yè)人士 2015-02-16